“Creo que este es uno de los antibióticos más potentes que se han descubierto hasta la fecha”, afirma el bioingeniero James J. Collins en ‘The Guardian’ cuando habla de la halicina. Las propiedades antibióticas de esta molécula se han descubierto gracias a un proceso de deep learning que pretendía sentar las bases para realizar, precisamente, este tipo de descubrimientos.
El nombre de la halicina (halicin) proviene de HAL9000, la inteligencia artificial de ‘2001: una odisea en el espacio’ (1968). Si es interesante es por la forma en que se ha descubierto mediante deep learning y por su capacidad para derrotar a bacterias como Clostridium difficile, Acinetobacter baumannii, Mycobacterium tuberculosis o algunas cepas de Enterobacteriaceae.
La halicina no es una molécula nueva
La halicina no es nueva para nosotros. Llevábamos años investigando a la hasta ahora molécula SU3327 para tratar la diabetes, sin éxito. Sin embargo, la red neuronal diseñada por Regina Barzilay y James J. Collins supo ver una conexión entre características de la SU3327 y la inhibición del crecimiento de la E.coli (abajo) por otras moléculas. La idea tras el experimento es como sigue.
Desde hace décadas los investigadores enfrentan diferentes compuestos de crecimiento bacteriano en simuladores virtuales (in silico), placas petri (in vitro), animales (in vivo) y, finalmente, seres humanos. Con frecuencia con resultados nulos en el objetivo a tratar, ya que la mayoría de los fármacos testados no acaban siendo efectivos para esa tarea concreta.
Pero ¿y si fuesen útiles para algo distinto? Para ello los datos de estos descubrimientos negativos se almacenan junto a toda clase de factores tales como la forma de la molécula o su comportamiento, por poner dos. Esta base de datos por sí sola no tiene por qué aportar demasiada información a los investigadores. Básicamente es un listado molécula por molécula con columnas repletas de factores de difícil conceptualización.
Es como tener los análisis clínicos impresos de toda la población española y pedir a un médico que eche un vistazo y nos diga qué tal vamos. Nos hace falta un análisis en profundidad, nuevas herramientas y un objetivo.
¿Cómo se ha descubierto este nuevo antibiótico?
Es aquí donde entra el algoritmo de deep learning al que se le pide encontrar un patrón entre fármacos previos y nuevas moléculas. ¿El objetivo? Que el nuevo fármaco ayude a inhibir el crecimiento de la E.Coli. Analizando 107 millones de compuestos (de una base de datos con 1.500 millones, volveremos a esto más adelante), se han descubierto 23 compuestos potencialmente antibióticos porque representan correlaciones con antibióticos que sabemos que funcionan.
Para entender el fenómeno, es como descubrir el potencial de bailar bien de una persona que nunca lo ha intentado solo porque come de forma parecida a los bailarines profesionales y le gustan las mismas películas. Todas estas correlaciones tan complejas de ver para los humanos son más sencillas para algoritmos de deep learning, capaces de localizar patrones escondidos.
Existe una gran cantidad de correlaciones que a los humanos nos cuesta mucho identificar pero que un algoritmo encuentra rápidamente en una base de datos dada. Pero, una vez obtenidos estos resultados enteramente virtuales, hay que verificarlos como ha ocurrido con la halicina. Si cumplen su objetivo, el algoritmo de deep learning ha hecho bien su trabajo. Si no, se seguirá iterando.
De momento, la halicina ha sido una de las moléculas probadas con cierto éxito in vitro y con modelos murinos in vivo. Además, ocho compuestos más destacan no solo por su (aún teórica) capacidad para inhibir el crecimiento de la E.Coli, sino porque no tienen la forma molecular que esperábamos. Igual estamos ante el nacimiento de una nueva rama de antibióticos de amplio espectro.
¿Por qué hacen falta máquinas para dar con antibióticos?
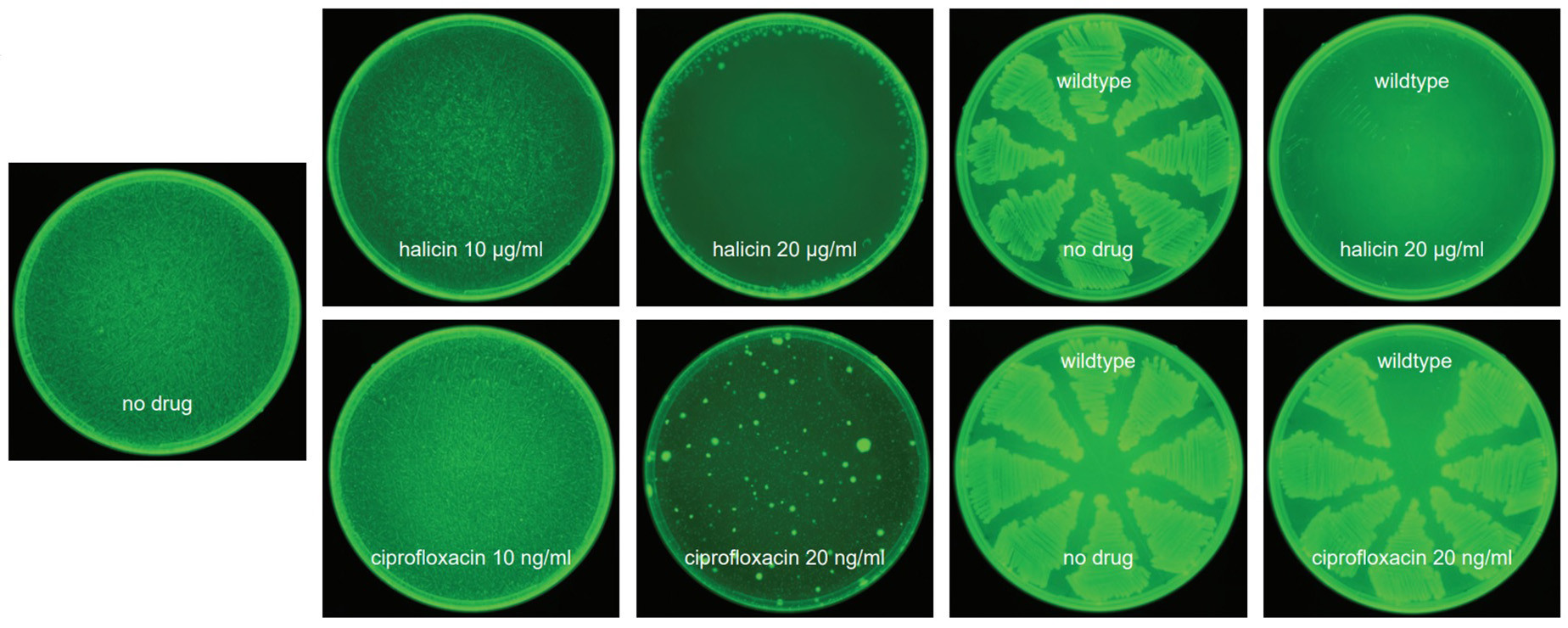
Aunque pueda parecer extraño, hace muchas décadas que no descubrimos nuevos grupos de antibióticos altamente eficaces y de amplio espectro. El descubrimiento de las penicilinas y sus variantes supuso un enorme hito en materia de salud. Pero es un hito que aún no se ha superado, ni siquiera con los carbapenem o las diferentes generaciones de cefalosporinas.
Cada vez tenemos más antibióticos, pero estos son de espectro más cerrado o reducido. Es decir, pueden usarse para menos tratamientos y están más contraindicados para un mayor número de casos. Y a esto se suma el aumento de la resistencia bacteriana a muchos de los antibióticos, reduciendo aún más su eficiencia y rango de aplicación. Hablamos de las llamadas superbacterias.
Las superbacterias no son bacterias grandes, pero sí más resistentes. Han mutado más rápido que nuestros fármacos, se han vuelto resistentes a muchos antibióticos y los han dejado obsoletos. Hace años que la OMS relacionó un mal uso de los antibacterianos con el auge de las superbacterias. Y de hecho la actual epidemia de coronavirus podría aumentar la resistencia de estas.
¿Puede el SARS-CoV-2 influir en las superbacterias?
Lo cierto es que sí, por un aumento en el uso de antibióticos, tanto racional como irracional. Empecemos por este último para recordar que los antibióticos no destruyen los virus, salvo contadísimas excepciones basadas en procesos intermedios. Un aumento de su consumo debido a la desinformación supone un problema para todos. Las bacterias ‘aprenden’ al acumular mutaciones.
Pero incluso con un uso racional de antibióticos, el SARS-CoV-2 puede contribuir a las superbacterias. Uno de los rasgos de la COVID-19 es el debilitamiento de las defensas del cuerpo, lo que abre la puerta a infecciones bacterianas que sí han de ser tratadas con antibióticos. Al aumentarlos, corremos el riesgo de contribuir a futuras superbacterias. Más uso de antibióticos, más superbacterias.
Más fuerza a los antibióticos in silico
Las pruebas farmacológicas in silico están en auge por varios motivos, pero el más importante es que ayudan a sesgar mucho el número de fármacos tratados. Lo hemos visto con el caso de Regina Barzilay y James J. Collins, que partían de una muestra de 2.335 moléculas. El algoritmo les devolvió primero 99, de las cuales 51 mostraron una inhibición del crecimiento de la bacteria E.Coli.
Es decir, el algoritmo redujo la muestra al 4% en la primera ronda. Imaginemos lo que habría supuesto en gasto haber probado todas las moléculas originales. Ese es el poder del deep learning.
A mediados de 2018 teorizamos sobre las posibles consecuencias de este tipo de análisis al hablar de un algoritmo que redescubrió la tabla periódica, y en menos de dos años tenemos un antibiótico. Eso sí, la halicina aún tiene que ser testada en personas, un proceso lento que suele requerir años o décadas.
En Nobbot | Tu ordenador también puede ser un arma en la lucha contra el coronavirus
Imágenes | CDC, CDC, Barzilay & Collins, NCI